Biologia molekularna, genetyka
Biologia molekularna, genetyka
Zróżnicowanie genetyczne oraz identyfikacja mikroorganizmów glebowych
(PCR, Real Time PCR, PCR DGGE, Biolog System)




Możliwe zastosowania systemu Biolog:
- identyfikacja mikroorganizmów z wykorzystaniem płytek GenIII
- analiza funkcjonalna wybranych szczepów – krzywe wzrostu, tempo rozkładu różnych substratów (do wyboru kilkaset różnych źródeł węgla i azotu)
- wpływ dodatków (antybiotyków, środków ochrony roślin, nawozów … ) na wzrost i tempo rozkładu substratów przez bakterie lub grzyby
- analiza bogatych gatunkowo próbek środowiskowych (gleba, ścieki, woda) pod kątem całościowego metabolizmu konsorcjów bakteryjnych (płytki ECO) lub grzybowych (FF)
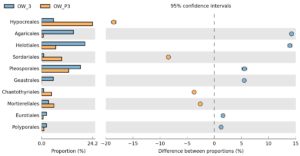
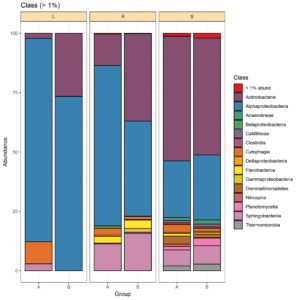
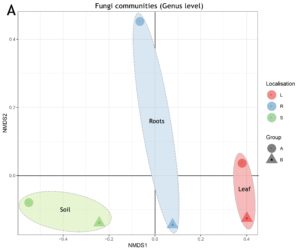
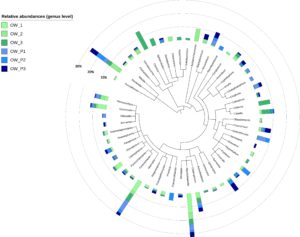
Metagenomika - analiza sekwencjonowania amplikonów
Metagenomika - analiza sekwencjonowania amplikonów
Analizy bioinformatyczne – sekwencjonowanie amplikonów 16S (bakterie) i ITS (grzyby)
- kompletna analiza od surowego pliku fastq
- opracowanie statystyczne, prezentacja graficzna
- alfa i beta bioróżnorodność
DGGE - PCR
DGGE - PCR
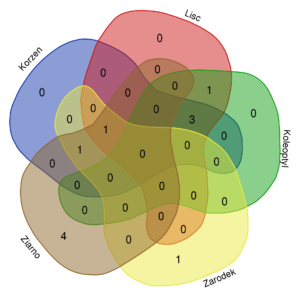
Ocena różnorodności mikrobiologicznej gleb w oparciu o technikę PCR DGGE

Bakterie symbiotyczne
Bakterie symbiotyczne
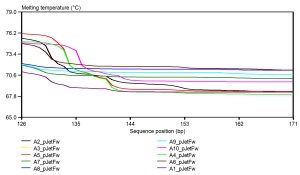
Badania nad właściwościami i praktycznym wykorzystaniu bakterii zdolnych do wiązania azotu atmosferycznego jak i bakterii symbiotycznych roślin bobowatych (NITRAGINA)



Ponadto:
- ocena różnych parametrów mikrobiologicznych, biochemicznych i ruchomych frakcji materii organicznej jako wskaźników żyzności i zdrowotności gleby oraz analiza wpływu różnych zabiegów agrotechnicznych na te wskaźniki,
- określenie zawartości glomalin w glebach Polski,
- ocena oddziaływania różnych zabiegów agrotechnicznych i systemów gospodarowania na zasiedlanie kłosów, ziarna i korzeni zbóż przez grzyby mykoryzy arbuskularnej (AM) oraz toksynotwórczych grzybów z rodzaju Fusarium występujących za ziarnie zbóż.
1) Wstępne analizy gleby:
- Oznaczanie suchej masy gleby.
- Pomiar pH i EC gleby.
- Oznaczenie całkowitej pojemności wodnej gleby.
2) Analizy środowiskowe gleby:
- Analiza aktywności enzymatycznej gleby:
- Dehydrogenaz;
- Fosfatazy kwaśnej i zasadowej.
- Dehydrogenaz;
- Oznaczanie biomasy (C i N) w próbkach glebowych metodą fumigacji-ekstrakcji.
- Analiza mikrobiologiczna próbek glebowych (metoda klasycznych wysiewów na płytki).
- Analiza ruchomych frakcji węgla:
- Analiza zawartości węgla organicznego ekstrahowanego zimną i gorącą wodą;
- Oznaczanie zawartości POM w glebie.
- Oznaczanie zawartości glomalin ogólnych oraz łatwo-ekstrahowanych w glebie.
3) Analizy z wykorzystaniem technik molekularnych:
- Izolacja DNA glebowego.
- Łańcuchowa reakcja polimerazy (PCR).
- Elektroforeza w żelu agarozowym.
- Elektroforeza w żelu z gradientem czynnika denaturującego (DGGE).
4) Analiza prób glebowych pod kątem zróżnicowania metabolicznego z użyciem Biolog System.
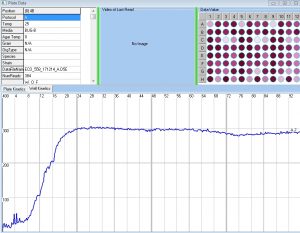
System Biolog – analiza metabolizmu oraz fenotypowanie mikroorganizmów
Aparatura wykorzystywana jest do analizy różnorodności funkcjonalnej mikroorganizmów w próbkach środowiskowych (np. gleby użytkowane rolniczo, gleby skażone, biopreparaty).

Liofilizator FreeZone Triad System

Kompletny system liofilizacyjny łączący w sobie kaskadowy liofilizator stołowy oraz moduł do liofilizacji materiału w penicylinówkach i zamykania pod próżnią. Aparat umożliwia przeprowadzenie kompletnego procesu liofilizacji, poczynając od wstępnego zamrażania materiału w penicylinówkach (lub luzem) do temperatury -75 °C, poprzez proces liofilizacji, aż do automatycznego zamknięcia penicylinówek w atmosferze panującej próżni.
Kaskadowy, wolnyod freonu system chłodzący pozwala na chłodzenie spirali kolektora do temperatury -85 °C. Liofilizację materiału prowadzić można na półkach albo w naczyniach liofilizacyjnych podłączonych do czterech zaworów umiejscowionych na
lewej ścianie bocznej aparatu.
Platforma bioinformatyczna
Podzespoły takie jak:
- 24-rdzeniowy procesor (48 wątkowy) z 64 MB pamięci podręcznej,
- 128 GB pamięci RAM
- Dysk twardy SSD
Wraz z oprogramowaniem:
- BioNumerics Fingerprint Data
- BioNumerics Sequence Data
- Bionumerics Genome Analysis Tools
- BioRad QuantityOne
- Pakiet R (RStudio, wtyczki: m.in phyloseq, dada2, vegan, ggplot2)
- mothur (v. 1.40.0 i nowsze)
- Qiime2 (wersja 2018.11 i nowsze)
- Seed2
- Unipro UGENE
- i wiele innych
umożliwiają analizę i obróbkę surowych danych uzyskanych po sekwencjonowaniu następnej generacji (NGS: 16S, ITS, metatranskryptomika, RNA-seq, WGS) oraz przeprowadzanie skomplikowanych obliczeń w analizie kinetyki wzrostu z wykorzystaniem sytemu Biolog (EcoPlate, FF, GenIII, PM).
Homogenizator

Aparat ma zastosowanie do izolacji DNA z prób o różnym pochodzeniu (bakterie, grzyby, gleba, roślina), co pozwala na szybką i precyzyjną metodę pozyskania materiału genetycznego o bardzo wysokiej jakości i wysokiej wydajności izolacji. Homogenizator jest stosowany w badaniach z zakresu biologii molekularnej.
Spektrofotometr do DNA i RNA

Jest to urządzenie umożliwiające precyzyjną ocenę ilości wyizolowanego materiału genetycznego (DNA, RNA). Aparat wykorzystywany jest w badaniach z zakresu biologii molekularnej. Na tej podstawie można precyzyjnie ocenić zarówno ilość, jak i jakość DNA wyizolowanego z gleby (wskaźnik jakości środowiska glebowego), a także DNA wyizolowanego z bakterii i grzybów.
Komora do namnażania i przechowywania drobnoustrojów

Urządzenie do namnażania różnych gatunków kultur bakteryjnych i grzybowych w określonych warunkach wzrostu (temperatura, czas, naświetlenie). Komora posiada również możliwość instalacji oświetlenia fotosyntetycznego i/lub UV (sterylność prowadzenia badań). Ponadto dysponuje również zaawansowanymi funkcjami kontroli prowadzonych eksperymentów i możliwościami automatyzacji doświadczeń, co znacznie podwyższa jakość i sprawność prowadzonych badań.
Universal Mutation Detection System

Universal Mutation Detection System jest zestawem umożliwiającym wykonanie stosunkowo łatwej i precyzyjnej identyfikacji gatunkowej mikroorganizmów dominujących w danym siedlisku. System ten wykorzystuje metodę PCR-DGGE (ang. Denaturant Gradient Gel Electrophoresis). Jest on także używany w badaniach z zakresu biologii molekularnej i oceny zróżnicowania genetycznego drobnoustrojów pochodzących z różnych środowisk.
Realtime PCR – QPCR

Bioanalizator wraz z systemem QPCR – system bazujący na ilościowej reakcji łańcuchowej polimerazy DNA (qPCR, z ang. quantitative PCR; nazywana też PCR w czasie rzeczywistym). System ten jest wykorzystywany do identyfikacji gatunkowej bakterii i grzybów w badaniach z zakresu biologii molekularnej i genetyce.
Inkubator z wytrząsaniem

Inkubator z wytrząsaniem – aparatura ta jest wykorzystywana do namnażania hodowli określonych drobnoustrojów (bakterii, grzybów) na określonych podłożach wzrostowych.
System do przygotowywania ultraczystej wody

System do przygotowywania ultraczystej wody. Stosowanie do analiz biologicznych i chemicznych ultraczystej wody o określonych parametrach jest podstawą do uzyskania wiarygodnych i precyzyjnych wyników pomiarów.
Komora laminarna

Urządzenie wykorzystywane jest do pracy laboratoryjnej (mikrobiologicznej, genetycznej) w warunkach sterylnych.
Komora laminarna II klasy bezpieczeństwa używana jest w laboratorium biologii molekularnej (izolacja DNA, przygotowywanie reakcji PCR) oraz w pracy mikrobiologicznej (przeszczepianie izolatów bakteryjnych