W dniu 13.12.2017 mgr Jarosław Grządziel wygłosił w IUNG-PIB prezentację podsumowującą realizację półrocznego projektu pt. „Opracowanie wzorców molekularnych do szybkiej identyfikacji dominujących bakterii zasiedlających różne środowiska glebowe, z wykorzystaniem techniki elektroforezy w gradiencie środka denaturującego (DGGE)”.
Projekt realizowany był w ramach Dotacji dla Młodych Naukowców 2017, finansowanej przez Ministerstwo Nauki i Szkolnictwa Wyższego.
Kierownik projektu: mgr Jarosław Grządziel,
Opiekun: dr Anna Gałązka
Badanie przeprowadzono z wykorzystaniem dwóch obiektów doświadczalnych. Jednym z nich były doświadczalne mikropoletka należące do Instytutu, na które składa się osiem różnych gleb, charakterystycznych dla Polski. Drugim obiektem była gleba skażona ropą naftową pobrana spod aktywnego szybu naftowego w Węglówce.


Pierwszym etapem badań była izolacja całkowitego DNA pochodzącego ze społeczności mikroorganizmów glebowych. Następnie przeprowadzono szereg reakcji PCR w celu dobrania optymalnych warunków powielenia hiperzmiennego fragmentu V3 genu 16S rRNA.
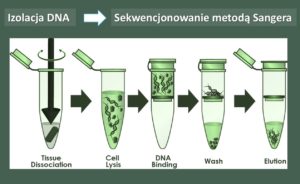
Otrzymane krótkie fragmenty DNA rozdzielono w żelu poliakrylamidowym z zastosowaniem metody DGGE, która umożliwiła zobrazowanie różnorodności mikrobiologicznej badanych siedlisk, w postaci prążków na żelu.
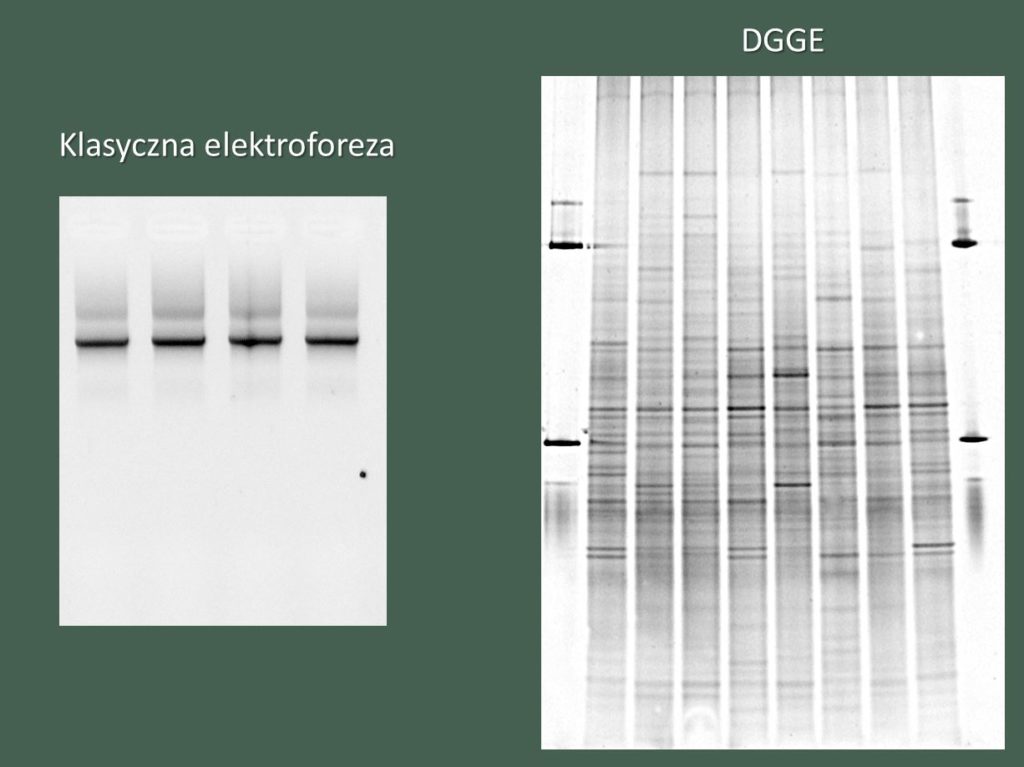
Do dalszych analiz wybrano siedem prążków pochodzących z rozdziału DNA wyizolowanego z gleb należących do doświadczenia mikropoletkowego IUNG oraz osiem prążków z gleby skażonej ropą. Prążki te wycięto z żelu za pomocą sterylnej końcówki do pipety, umieszczono w sterylnej probówce oraz dodano 100 ul buforu TE. Zawieszone w buforze prążki inkubowano przez 16 godzin w 4°C, następnie wytrząsano przy 600 rpm w 50°C, oraz odwirowano przy 12 000 x rcf. Eluat stanowił matrycę do reakcji PCR w celu reamplifikacji krótkich fragmentów DNA otrzymanych po wycięciu z żelu.

Kolejnym etapem było przeprowadzenie klonowania otrzymanych produktów PCR z wykorzystaniem szczepu Escherichia coli TOP10 oraz wektora pJET 1.2. Bakterie transformowane wektorem zawierającym przygotowany wcześniej produkt PCR zostały wysiane na podłożu LB z dodatkiem ampicyliny, w celu selekcji pozytywnej mutantów. Z każdej płytki wybrano po 10 losowych kolonii, z których wykonywano reakcje PCR z zastosowaniem tych samych starterów, które zostały użyte w pierwszym etapie. Produkty te następnie poddane zostały ponownej elektroforezie metodą DGGE w celu potwierdzenia, czy otrzymane produkty odpowiadają wysokością fragmentom DNA z których zostały wyizolowane.


Ostatnim etapem było sekwencjonowanie metodą Sangera otrzymanych produktów oraz porównanie uzyskanych sekwencji z dostępnymi bazami danych.
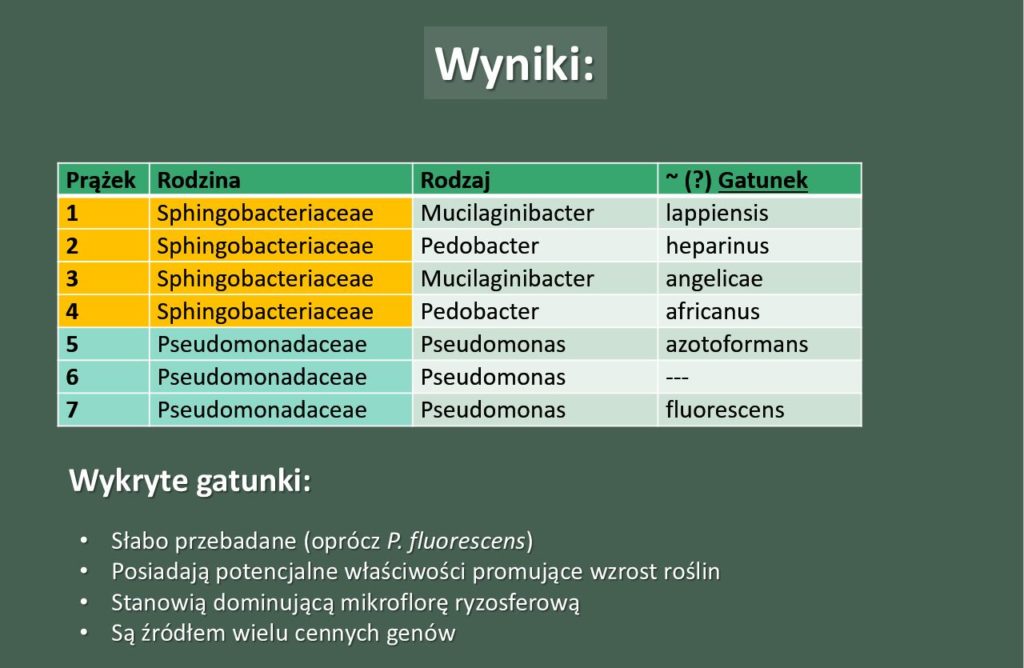
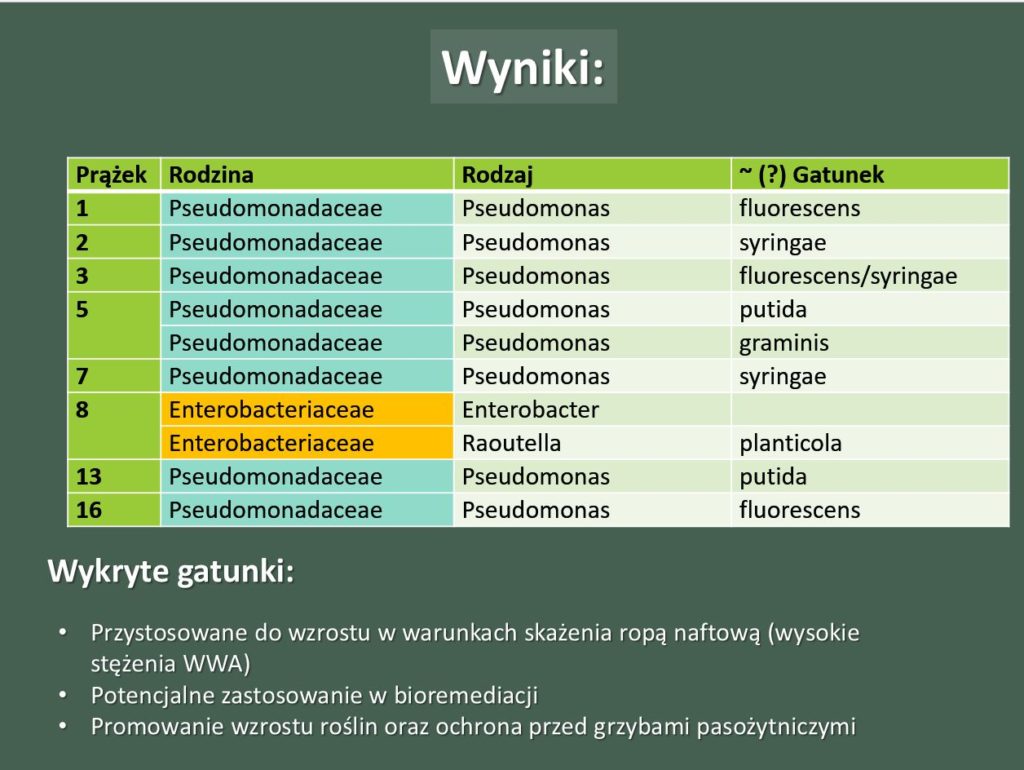
Przeprowadzone analizy potwierdziły, że dominującymi bakteriami w glebie są gatunki niehodowalne tradycyjnymi metodami. Należą one głównie do rodzajów Mucilaginibacter, Pedobacter, Pseudomonas w przypadku gleb uprawowych oraz Pseudomonas, Enterobacter, Raoutella, w przypadku gleby skażonej ropą. Wśród sklasyfikowanych, hodowalnych bakterii znalazły się zarówno bakterie potencjalnie przyspieszające wzrost roślin, takie jak Pseudomonas azotoformans, które mogą w przyszłości posłużyć do produkcji szczepionek bakteryjnych oraz bardzo groźne patogeny roślin np. Pseudomonas syringae, których obecność w glebach skażonych należy brać pod uwagę podczas prób rekultywacji gleb zdegradowanych.
Wyniki projektu były prezentowane m.in.:
• Na międzynarodowej konferencji:
- Metagenomic approach to investigate the microbiota of different soil types. 7th International Weigl Conference, 26-29.09.2017, Lwów, Ukraina.
- Identification of dominant bacteria in different types of soil using PCR-DGGE molecular standards. 7th International Weigl Conference, 26-29.09.2017, Lwów, Ukraina.
• Na krajowej konferencji:
- Opracowanie wzorców molekularnych do szybkiej identyfikacji zmian populacji mikroorganizmów w środowiskach glebowych z wykorzystaniem metody DGGE. 51 Ogólnopolska Konferencja Naukowa, „Mikrobiologia środowiskowa szansą bezpiecznego życia i postępu biotechnologicznego”, 5-8.09.2017, Toruń-Ciechocinek.
JGrzadziel_Poster_Lwow